CRISPR diagnostics of phytopathogens: first steps and prospects
CRISPR diagnostics of phytopathogens: first steps and prospects
Abstract
The use of CRISPR/Cas-nucleases for nucleic acid detection (CRISPR-diagnostics) has been actively developed since 2017-2018, after the first diagnostic platforms – DETECTR and SHERLOCK – emerged. CRISPR diagnostics is based on the pairing of isothermal nucleic acid amplification methods with selective recognition of target amplicons by Cas-nucleases. Since isothermal amplification and CRISPR/Cas detection do not require sophisticated equipment and the results can be evaluated by non-instrumental methods, this approach is promising for non-laboratory diagnostics, including field DNA diagnostics of agricultural plant diseases. This review briefly discusses the basic principles of CRISPR/Cas detection and isothermal nucleic acid amplification methods used in CRISPR diagnostics today. Examples of their coupling for the identification of different phytopathogens under in vitro and out-of-laboratory conditions and prospects for further development of CRISPR diagnostics of phytopathogens are also reviewed.
1. Введение
CRISPR-диагностика – направление в ДНК-диагностике, нацеленное на создание биосенсорных платформ для лабораторной и внелабораторной (формат «point-of-care testing» и «point-of-need testing» ) диагностики заболеваний человека, сельскохозяйственных животных и растений, в первую очередь инфекционных. Термин «CRISPR-диагностика» (англ. CRISPR diagnostics) был впервые введён в научный лексикон в 2021 г. в ряде публикаций в ведущих научно-исследовательских журналах (таких как Nature и Science) , , как новое направление в ДНК-диагностике.
Сегодня «золотым стандартом» диагностики инфекционных заболеваний является метод полимеразной цепной реакции (ПЦР), часто в формате ПЦР в реальном времени, что позволяет проводить количественную детекцию патогенных микроорганизмов и вирусов. Начиная с 1988 г., когда была открыта и внедрена в практику термостабильная ДНК-полимераза из Thermus aquaticus (Taq-полимераза), ПЦР-анализ начал своё широкое распространение, создав основу для развития нового направления – ДНК-диагностики инфекционных заболеваний, в том числе и ДНК-диагностики заболеваний сельскохозяйственных растений . Благодаря своей чувствительности и селективности, ПЦР быстро стал частью арсенала методов, рекомендованных для проведения фитосанитарного контроля и мониторинга состояния агрокультур .
Однако для выполнения ПЦР необходимы лабораторные условия, включая наличие квалифицированного персонала и достаточно сложных приборов, обеспечивающих циклический процесс амплификации и оптическую детекцию сигнала. Всё это затрудняет использование ПЦР как основы для развития полевой ДНК-диагностики фитопатогенов. Для успешного развития полевых тест-систем должны использоваться технологии детекции, характеризующиеся компактностью, низкой энергоемкостью, простотой использования, которые при этом позволяли бы оперативно (в течение 1-2 часов) получать результаты и интерпретировать их с помощью простого и недорогого оборудования. Хотя иммунохимические тесты отвечают этим требованиям, они характеризуются, как правило, невысокой чувствительностью и, часто, недостаточной селективностью. В настоящее время наиболее перспективной технологией для создания высокочувствительных и селективных тест-систем для полевой диагностики фитопатогенов представляется использование CRISPR/Cas-нуклеаз в сочетании с изотермической амплификацией нуклеиновых кислот .
2. Технологические основы CRISPR/Cas-диагностики
В основу CRISPR-диагностики положена способность CRISPR/Cas-нуклеаз приобретать так называемую коллатеральную активность (или trans-активность) после узнавания комплексом нуклеазы и нРНК (направляющая РНК; англ. guide RNA) ДНК- или РНК-мишени , . Узнавание происходит путём формирования дуплекса между участком нРНК («спейсер») длиной 20-30 нуклеотидов с комплементарным ему участком («протоспейсер») ДНК- или РНК-мишени. Появление коллатеральной активности, указывающие на присутствие мишени, детектируется чаще всего по измерению интенсивности флуоресценции, которое вызывается расщеплением активированной Cas-нуклеазой коротких ДНК- или РНК-олигонуклеотидов – «репортёров». «Репортёры» несут на 5′- и 3′-концах флуорофор (как правило, флуоресцеин) и молекулу, способную поглощать испускаемые флуорофором фотоны («гаситель»). При их расхождении в пространстве при деградации «репортёра» Cas-нуклеазой, «гаситель» теряет способность эффективно поглощать испускаемые фотоны, что приводит к значительному (в десятки и сотни раз) возрастанию интенсивности флуоресценции. Возрастание флуоресценции может быть определено как инструментальными методами с использованием флуориметра, так и визуально, невооружённым глазом (неинструментальная детекция), при освещении тест-пробы синим (длина волны 400-500 нм) светом по изменению её окраски (колориметрический метод) . Также возможна детекция иммунохимическим методом с помощью коммерческих тест-полосок – для этого «репортёры» должны нести на концах молекулы флуоресцеина и биотина .
К наиболее широко используемым в диагностических платформах CRISPR/Cas-нуклеазам относятся Cas13a, Cas12a и Cas12b, при этом мишенью для первой является молекула РНК, в то время как нуклеазы Cas12 распознают в качестве мишени как двухнитевые, так и однонитевые последовательности ДНК . Также в качестве детектирующих нуклеаз могут быть использованы Cas9, Cas3 и Cas12f, однако их применение в тест-системах либо ограничивается необходимостью использования сложных рибопротеиновых комплексов, требующих раздельной экспрессии и очистки (как в случае Cas3), либо не имеет преимуществ перед более распространенными нуклеазами. На рисунке 1 схематически показан принцип детекции полинуклеотидной молекулы с использованием CRISPR/Cas-нуклеаз, включающий связывание направляющей РНК с ферментом с последующим распознаванием целевой последовательности .
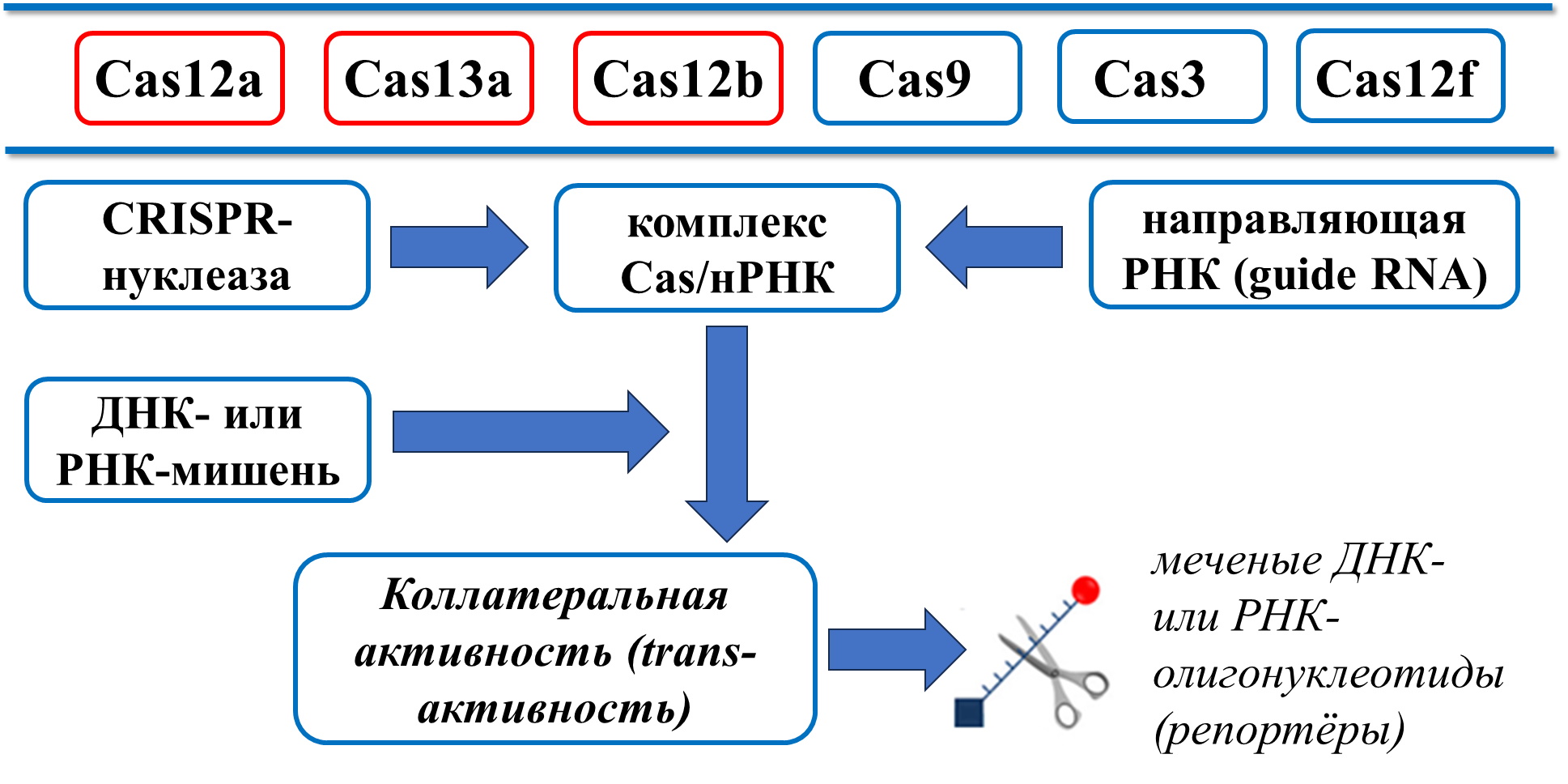
Рисунок 1 - Схематическая иллюстрация метода CRISPR/Cas-детекции. Красным отмечены Cas-нуклеазы, наиболее часто используемые в CRISPR-диагностике
Таблица 1 - Методы изотермической амплификации, используемые в комбинации с CRISPR/Cas-детекцией ампликонов
Метод амплификации | Мишень | Продукт | Температура (℃) | Время (мин) |
LAMP (loop mediated amplification) | ДНК | ДНК-ампликоны | 60-65 | 60 |
RPA (recombinase polymerase amplification) | ДНК | ДНК-ампликоны | 37 | 20-30 |
NASBA (nucleic acid sequence based amplification) | РНК | РНК-ампликоны | 41 | 90 |
К недостаткам тестирования с помощью сочетания CRISPR/Cas-детекции с изотермической амплификацией можно отнести необходимость проведения реакций в два этапа с переносом ампликонов в новую пробирку, содержащую смесь Cas-нуклеазы с направляющей РНК и молекулами-репортёрами, а также чувствительность основных ферментов к повышенной температуре и циклам замораживания-оттаивания, что создает сложности при транспортировке и хранении тест-систем в полевых условиях.
Для решения указанных выше проблем в настоящее время сформировалось несколько основных трендов в полевой диагностике, основанной на CRISPR/Cas-детекции. В первую очередь при разработке новых тест-систем исследователи стремятся к осуществлению диагностики в формате одной пробирки . В том случае, если буферные системы изотермической амплификации и Cas-детекции оказываются несовместимыми, применяют физическое разделение компонентов за счет размещения одной из реакций на крышке пробирки в виде капли. Однако данный подход затруднителен в случае, когда амплификация ДНК происходит с помощью метода LAMP, который проводится при достаточно высоких температурах. Для сопряжения LAMP с CRISPR/Cas-детекцией в формате одной тест-пробирки было предложено использовать термостабильную Cas-нуклеазу Cas12b, которая может проявлять коллатеральную активность при температурах вплоть до 67℃ . Также проводятся исследования возможности использования реагентных смесей (включая ферменты для изотермической амплификации и Cas-нуклеазы) в лиофилизированном виде (что в значительной степени решает проблемы хранения и транспортировки реагентов при комнатных температурах) и интегрирования изотермической амплификации и CRISPR/Cas-детекции ампликонов с микрофлюидными системами, упрощающими проведение CRISPR-диагностики во внелабораторных условиях.
3. Современное состояние CRISPR-диагностики фитопатогенов
Несмотря на стремительный рост числа исследований в области CRISPR-диагностики за последние 5 лет, примеры её использования для детекции фитопатогенов немногочисленны, поскольку подавляющая часть работ посвящена выявлению возбудителей инфекционных заболеваний человека и животных, либо технологическим аспектам разрабатываемых биосенсорных платформ с использованием CRISPR/Cas-нуклеаз. Одним из примеров биосенсорных платформ на основе CRISPR/Cas-нуклеаз для идентификации фитопатогенов является работа Y. Guo с сотр. . Комбинация RPA и детекции ампликонов с помощью Cas12a-нуклеазы позволила создать тест-систему для идентификации Phytophthora ramorum (фитопатогенный оомицет, вызывающий фитофтороз древесных и кустарниковых культур) с чувствительностью на уровне 100 пг геномной ДНК в исходной пробе за 25 минут при 37℃. Результаты теста могли быть оценены как с помощью планшетного ридера, так и визуально, при освещении пробирок светом с длиной волны 470 нм.
В работе Kang с сотр. для диагностики патогенных грибов Magnaporthe oryzae Triticum (инфекция, вызывающая пирикуляриоз – ожог колоса пшеницы) использовалась как изотермическая амплификация LAMP, так и сочетание CRISPR/Cas-нуклеазы Cas12a с RPA. Актуальность теста на данное заболевание обусловлена тем, что заражение не вызывает визуальных симптомов у пшеницы до стадии колошения, когда применение фунгицидов уже неэффективно. Амплификация целевого участка генома Magnaporthe oryzae Triticum с помощью LAMP позволила однозначно дифференцировать его от родственного патотипа Magnaporthe oryzae Oryzae, поражающего рис. Однако более перспективным подходом для создания полевого теста была признана комбинация Cas12a с изотермической амплификацией RPA. Для визуальной оценки результатов проводился второй раунд RPA с праймерами, мечеными FAM (левый праймер) и биотином (правый праймер), целевая последовательность для которых находилась в одноцепочечной ДНК, добавляемой в реакцию в качестве неспецифической мишени для нуклеазы Cas12a. По окончании реакции результаты оценивались с помощью коммерческих иммунохроматографических тест-кассет. В том случае, если в исходной пробе находились целевые ампликоны, активированная нуклеаза Cas12a деградировала неспецифическую мишень и не синтезировался продукт с двумя меченым праймерами, что приводило к появлению одной полоски на тест-кассете. При отсутствии в пробе геномной ДНК Magnaporthe oryzae Triticum в процессе второй RPA мишень для меченых праймеров остается интактной и на тесте проявляются две полоски. Авторами также показано, что подобный подход позволил не только повысить чувствительность на порядок (обнаружение 0.001 мкг на мкл-1 вместо 0.01 мкг на мкл-1 геномной ДНК) по сравнению с обычной ПЦР, но и сократить срок обнаружения инокулированной инфекции до 2-х дней вместо 4-х .
Также для идентификации патогенов может быть адаптирована платформа Bio-SCAN , в которой используются нРНК и меченая биотином рекомбинантная версия мутантной CRISPR-нуклеазы Cas9, лишённая нуклеазной активности (bio-dCas9). Меченый FAM (6-карбоксифлуоресцеин) ампликон, получаемый в результате RT-RPA (амплификация, сопряжённая с обратной транскрипцией), дополняется биотиновой меткой только в том случае, если он целевой и может быть узнан комплексом нРНК/ bio-dCas9. Селективное связывание bio-dCas9 с целевыми ампликонами детектируется с помощью коммерчески доступных иммунохроматографических тест-полосок (содержащих зону с иммобилизованным стрептавидином). В работе Sanchez с сотр. описывается применение технологии Bio-SCAN для выявления устойчивых аллелей пшеницы, синтетических мутантов риса, ГМО (по наличию промоторов, используемых для экспрессии трансгенов), патогенных вирусов (вирус желтого скручивания листьев томата (TYLCV), вирус табачной мозаики (TMV) и картофельный вирус Y (PVY)), грибковой инфекции Puccinia striiformis f. sp. tritici и Magnaporthe oryzae Triticum (MoT) у пшеницы, а также бактериальные патогены Pseudomonas и Agrobacterium tumefaciens у N. benthamiana. Относительная простота, универсальность, высокая специфичность и чувствительность делают данную платформу перспективным инструментом полевого скрининга при селекции сельскохозяйственных культур, трансгенезе растений, синтетической эволюции растений и для быстрого выявления фитопатогенов.
Сочетание визуализации результатов с помощью иммунохроматографических тест-полосок и RPA/Cas12a-детекции использовалось и для идентификации возбудителя пирикуляриоза (ожога) риса Magnaporthe oryzae . Важным дополнением тест-системы в данном случае являлась использованная авторами методика выделения геномной ДНК из рисовых листьев и мицелия с помощью полосок фильтровальной бумаги, осуществляемая при комнатной температуре и без этапов центрифугирования .
В работе Aman с сотр. было показано успешное применение комбинации RT-RPA и CRISPR/Cas-нуклеазы Cas12a для идентификации вирусных инфекций растений. Это позволило создать тест-системы для детекции вируса табачной мозаики (TMV) и вирусов картофеля X и Y (PVX и PVY). При этом удалось совместить стадии обратной транскрипции, амплификации и CRISPR/Cas-детекции. Вся процедура детекции занимала 20 мин и проходила при 42°С. Показано, что в таком формате тестирования возможно обнаружение вирусной РНК в пикомолярных концентрациях, однако увеличение времени инкубации может снизить этот показатель до фемтомолярного уровня. Также стоит отметить, что для визуализации результатов в работе была показана возможность успешного использования простого и компактного прибора оптической детекции (стоимостью 35 долларов США), что делает предложенные тест-системы полностью отвечающими требованиям полевой диагностики.
На примере диагностики наиболее распространенных вирусных патогенов яблони, а именно вируса некротической мозаики (ApNMV), вируса ямчатости древесины (ASPV), вируса бороздчатости древесины (ASGV), вируса хлоротической пятнистости листьев (ACLSV) яблони и вироида рубцовой кожицы яблони (ASSVd) была показана возможность мультиплексного анализа в формате CRISPR-детекции . Платформа CRISPR/Cas12a-RT-RPA продемонстрировала чувствительность, сопоставимую с RT-qPCR, при этом пределы обнаружения достигали 250 копий вируса на реакцию для ASPV и ASGV и 2500 копий для остальных. Однако по сравнению со стандартным тестом с помощью RT-qPCR этот протокол был более быстрым и простым и требовал не более часа с момента сбора листьев. Такое сокращение времени анализа достигалось за счет упрощения процедуры выделения РНК (гомогенизация листьев в щелочном растворе полиэтиленгликоля) и использования неочищенного экстракта для RT-RPA, а также применения методики визуализации результатов невооруженным глазом благодаря конъюгированным с олигонуклеотидами золотым наночастицам.
В 2024 году коллективом авторов данного обзора была опубликована статья, описывающая успешный опыт по применению технологии DETECTR для детекции Dickeya solani — опасного фитопатогена, вызывающего заболевание картофеля, известное как “черная ножка”, с пределом обнаружения 1 копия бактериального генома на реакцию амплификации . При этом было показано, что даже в случае, когда изотермическая амплификация (в данном случае использовался метод RPA) не обладает необходимой селективностью для специфической детекции D. solani, последующая детекция целевых ампликонов с помощью CRISPR-нуклеазы Cas12a обеспечивает требуемую специфичность детекции для системы в целом .
Для другого бактериального патогена – Clavibacter sepedonicus, вызывающего кольцевую гниль картофеля, этим же коллективом авторов был разработан метод детекции, основанный на сочетании активности CRISPR-нуклеазы Cas13a с методом изотермической амплификацией NASBA . В формате анализа с проведением амплификации и Cas-детекции в отдельных пробирках, предел обнаружения составил 1000 копий целевой 16S рРНК на реакцию NASBA (одна бактериальная клетка содержит около 10 тыс. копий 16S рРНК) или около 24 колониеобразующих единиц (КОЕ) C. sepedonicus на 1 г ткани клубня картофеля. В формате "one-pot testing" (тестирование в одной пробирке) чувствительность понижалась до 10 тыс. копий 16S рРНК или около 100 КОЕ на 1 г ткани клубня. Тестирование может быть выполнено как с использованием флуориметра (инструментальная детекция), так и визуально по изменению цвета пробы при её освещении синим светом (неинструментальная детекция) . При этом общее время анализа не превышало 2 ч.
4. Заключение
Разработанные к настоящему времени платформы идентификации растительных инфекций продемонстрировали высокую чувствительность и селективность детекции и полное совпадение результатов определения фитопатогенов с результатами, полученными методом ПЦР, которая является «золотым стандартом» современной ДНК-диагностики. Однако до настоящего времени эффективность таких платформ была продемонстрирована исключительно в лабораторных условиях. Очевидно, что перспективы внедрения в сельскохозяйственную практику биосенсорных платформ для ДНК-диагностики фитопатогенов на основе CRISPR/Cas-нуклеаз, как предложенных к настоящему времени, так и разработанных в будущем, будут зависеть от результатов верификации их эффективности в условиях использования на агропредприятиях.
